導入事例
2018/05/25
[導入事例] 慶應義塾大学 中迫研究室様
NVIDIA TESLA P100-PCIe x8 ハイエンドサーバを導入頂きました。
研究内容

慶應義塾大学理工学部 中迫研究室 苙口友隆講師にお話を伺いました。
中迫研究室の紹介
慶應義塾大学 中迫研究室様では下記のような研究をされています。
▶ 蛋白質(ナノマシン)の構造解析、構造機能解析
▶ 蛋白質のダイナミクス測定と新たな測定技術開発
▶ X 線回折と分子動力学による蛋白質の水和構造解析
▶ SPring-8 および XFEL におけるX線散乱実験装置の開発
苙口先生の研究紹介
溶液中では蛋白質は軟らかく揺らいでおり、蛋白質機能にはこの軟らかさが重要であるという事に着目され、溶液散乱等の実験と、分子動力学シミュレーションや溶液理論を用いた計算を併用し、蛋白質の軟らかさと機能の関係を原子レベルで探る研究を行われています。

今回、NVIDIA TESLA P100 を8基搭載したハイエンドサーバと吐き出される膨大な量の計算データを管理する為の24ベイストレージサーバ等を導入されました。このシステムは研究室に設置されるとのことでしたので静音ラックをご提案し、OSのインストールから Amber や GROMACS、NAMD などのアプリケーションもインストールさせていただきました。
Q:今回導入されたシステムを使い、どのような研究をされるのかお聞かせください。
私達の研究室では、溶液中における蛋白質分子の機能的動きと、そのメカニズムを調べる研究を行っています。その目的のために、蛋白質分子とその周りの水分子も含めて10万個以上の原子からなる系の時間変化を、数百ナノ秒から数マイクロ秒にわたる時間スケールで、分子動力学(MD)シミュレーションにより追跡することを行っています。このような膨大な計算量を必要とするシミュレーションには、今回導入させて頂いたハイエンドサーバが必要不可欠です。

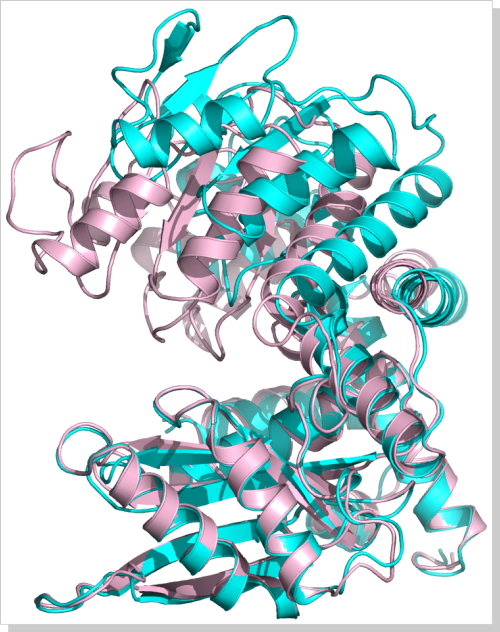
▼クリックすると拡大します。
図:ハイエンドサーバを用いたMDシミュレーションによって観察された、
蛋白質酵素分子が基質分子を捉える機能的動き
Q:いままではどのようなサーバをお使いでしたか。
これまでは、自前で計算サーバを保有したことはなく、東京大学情報基盤センターや自然科学研究機構計算科学研究センターの共用スーパーコンピューターを利用させて頂きながら、研究を進めてきました。これらの計算機を用いた成果は、幾つかの論文として発表しております。
導入システム

HPCT S24b-JBOD
SSD : 240GB x1
HDD : 4TB x23 RAID6
HPCT R424gs-8GP
GPU : NVIDIA TESLA P100-PCIe 16GB HBM2 x8
CPU : Intel Xeon E5-V4 22Core 2.2GHz x2
RAM : DDR4-2400 512GB
SSD : 240GB x1
HDD : 1TB x1
OS : CentOS6
Softwere : Amber, GROMACS, NAMD
HPCT S24b
CPU : Intel Xeon E5-V4 4Core 3.5GHz x1
RAM : DDR4-2400 32GB
SSD : 240GB x2
HDD : 8TB x24 RAID6 HotSpare2
HPCT SR45
防音特化型静音ラック
騒音減衰量:-24.3dB(A)
Q:導入後の感想を教えてください。
溶媒分子を含んだ蛋白質分子の系は、先述した通り極めて多くの原子からなる系であるため、その時間発展は複雑かつ確率的です。したがって、生物学的に意味がある蛋白質の動きを捉えるためには、複数の数マイクロ秒に及ぶシミュレーションにより、蛋白質の動きをより多くサンプリングする必要があります。今回導入させて頂いたハイエンドサーバにより、蛋白質の構造サンプリングをより多く行えるようになり、数百ナノ秒のシミュレーションでは捉えることができなかった蛋白質の動きが明らかになりつつあります。明らかにできる現象が増えることが期待できるので、これからの研究にワクワクしています。
最後に
苙口先生、ご多忙のところインタビューをさせていただき誠にありがとうございました。
ハードウェアサーポートをはじめ、理想の計算環境を実現できるよう、これからも全力でサポートさせていただきます。本日は、ありがとうございました。

▶ 苙口友隆 先生プロフィール
慶應義塾大学 理工学部 物理学科 基礎理工学専攻
中迫研究室ホームページ
http://www.phys.keio.ac.jp/guidance/labs/nakasako/nakasako-lab.html
研究テーマ
・生物物理学
・蛋白質構造解析
・分子シミュレーション
著書・発表論文・学会発表一覧
http://k-ris.keio.ac.jp/Profiles/210/0020924/profile.html