導入事例
2015/01/05
[導入事例] 慶應義塾大学 光武亜代理 先生
GPGPU4基搭載シミュレーション用計算機を2ノード導入頂きました。
研究内容
慶應義塾大学 光武亜代理 先生にお話しを伺いました。
研究内容の紹介
蛋白質の分子シミュレーションによる構造安定性とダイナミクスの研究
原子レベルで構造変化を追跡する際、分子シミュレーションは強力です。これまで、構造サンプリングを効率良く行う分子シミュレーションの手法の開発を行ってきました。今後もこの手法の開発を行っていこうと思っています。また、近年、長時間シミュレーションが可能になってきていて、動的な解析手法の開発が重要となっています。よって、得られた複雑な動きから遅い動き(モード)を抜き出す新規の動的解析手法の開発を行っています。そして蛋白質の遅い動きと機能の相関について調べたいと思っています。加えて、統計力学の液体論に基づく理論を用いて蛋白質のまわりの溶媒の効果を計算し、溶媒の寄与も含めた蛋白質の安定性についての研究も行っています。
導入システム
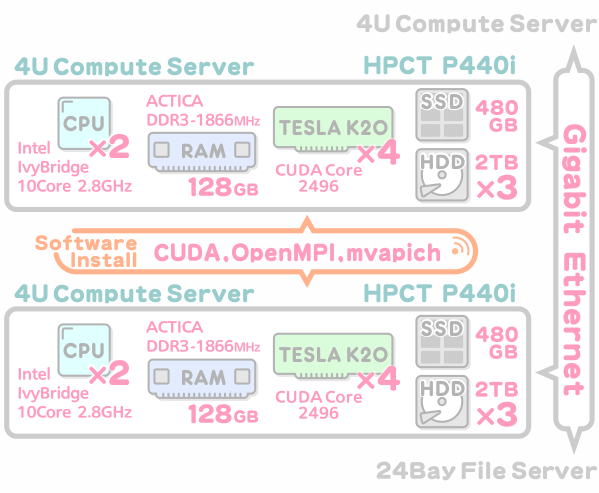

導入システムと使おうと思ったきっかけを教えてください。
タンパク質の分子シミュレーションを行うために、シミュレーションソフトウェアAMBERを主に利用しております。通常のCPUを使用するよりも、GPGPUを用いると高速に分子シミュレーションを実行できるため、GPGPUを搭載した計算機を導入しました。
今後の展望
今回は1ノードあたりTESLA K20を4基搭載した計算機を2台導入いたしましたが、今後のGPGPU単体の高性能化ならびに、ノード数を増やしていったときのGPU Clusterを視野にいれつつ、更なる高速化を行っていきたいと考えております。








